胚の染色体検査やERA(着床時期の検査)など、あらゆる場面で遺伝子検査によって判断することが増えています。
慢性子宮内膜炎の検査として、細胞を直接染める代わりに、子宮内の細菌の遺伝子を検出するようにする等、検査法も変わってきています。
遺伝子配列(DNA、RNA)を決定する方法は常に進化していますが、今、第3世代といえるシーケンサーが出てきました。
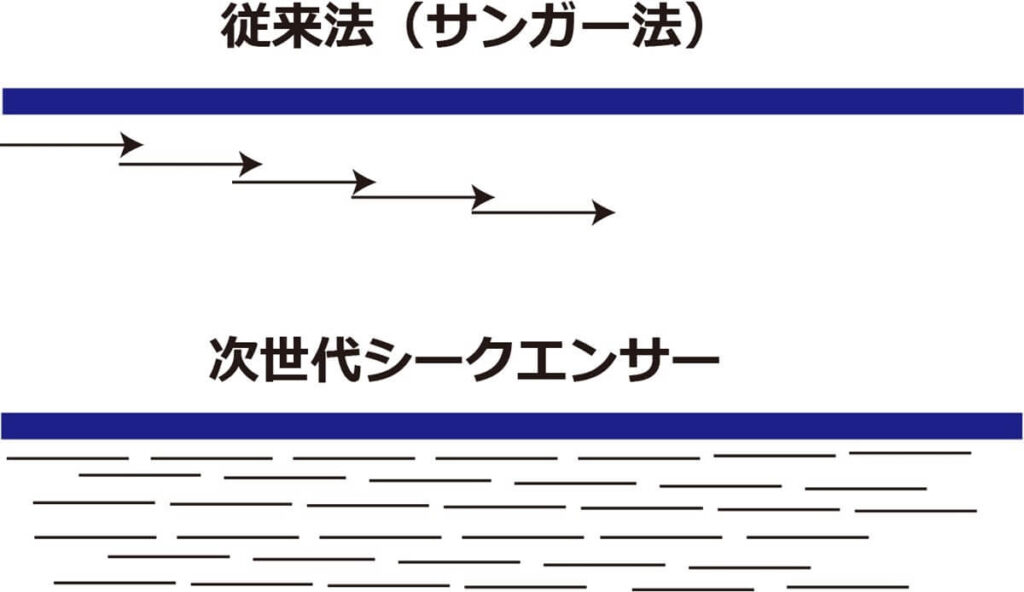
第1世代のDNAシーケンサーはサンガー法(Sanger sequencing)という方法に基づいています。
サンガー法は、1977年にフレデリック・サンガーによって開発され、DNAシーケンシングの分野で長らく主要な方法として使用されました。 サンガー法の基本原理は、DNAポリメラーゼを用いてDNA鎖を複製し、複製された鎖の中に特殊な蛍光マーカーを持つヌクレオチド(DNAを構成する成分)を挿入して、蛍光の強度に応じて順番にDNA鎖の塩基配列を特定します。
第2世代のシーケンサーは、今最もよく使われる「次世代シーケンサー」(NGS、Next-Generation Sequencing)と言われるものです。大量に高速に処理できるようになって驚きをもって迎えられたのがつい最近のことのよう。
開発は、2000年代初頭に始まり、その後急速に進展しました。 原理は、短い断片にして大量に増幅して読み取り、解析する方法です。
コンピューターやプリンターの進歩により可能になったと言えます。
とくにIlluminaシーケンサー(Solexa Sequencing)は、2005年に商業化され、今に至るまで各分野で使用されています。
【引用元】https://foodmicrob.com/dna-sequencers-princiiple/#次世代シークエンサー
そして、第3世代のシーケンサーと言われる中で最も注目されているのが「Nanopore sequencing」(ナノポアシーケンシング)です。
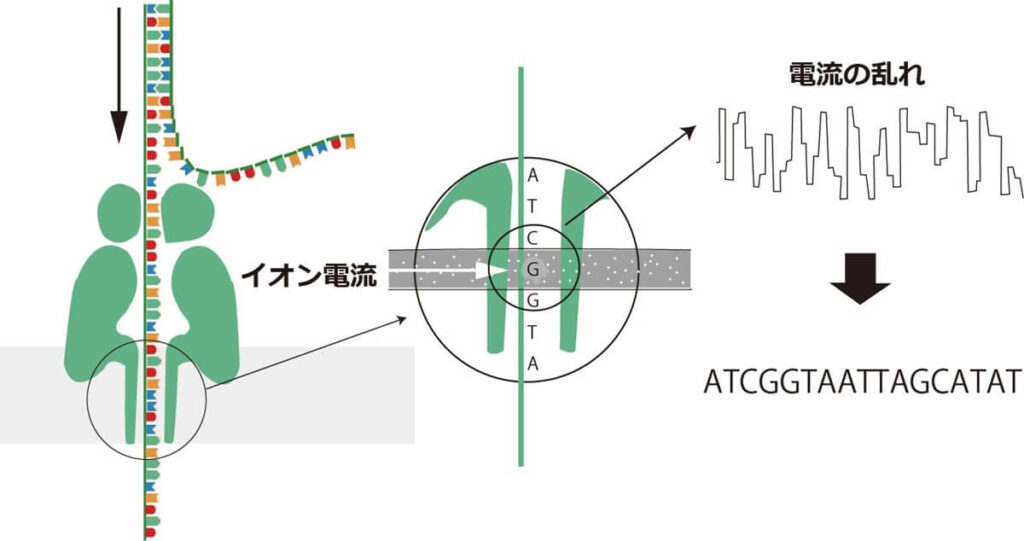
Oxford Nanopore Technologies(ONT)によって開発されたこの技術では、文字通りナノサイズの孔(ナノポア)を使用してDNA鎖を逐次読み取ります。
ナノサイズの小さな穴が空いている膜に電圧をかけることで、ナノポアにイオン電流を流すことができます。
このナノポアにDNAを通すと、通過するヌクレオチド(DNAの塩基の種類)毎に特徴的な電流の乱れが発生します。
この電流の変化を読み取ります。
【引用元】https://foodmicrob.com/dna-sequencers-princiiple/#原理
Nanopore sequencingは、多くの研究者や遺伝学者によって広く採用されていますが、その理由は、リアルタイムにデータ収集と解析ができるという点、長い配列を読み取ることが可能という点、さらに低コストである点です。
生命を扱う分野である生殖医療の分野での使用は慎重であるべきですが、今後さらに遺伝子の解析が簡単にできるようになることは、使い方を間違えなければ有益な技術だと思います。
【参考文献】
- The NEW ENGLAND JOURNAL of MEDICINE / Rapid Nanopore Sequencing–Based Screen for Aneuploidy in Reproductive Care / Aug 18, 2022
- National Library of Medicine / Nanopore Technology and Its Applications in Gene Sequencing / Jun 30, 2021
- National Library of Medicine / Rapid preimplantation genetic screening using a handheld, nanopore-based DNA sequencer / Oct, 2018
- National Library of Medicine / Third-generation sequencing: any future opportunities for PGT? / Feb, 2021
- Oxford Academic / Third-Generation Single-Molecule Sequencing for Preimplantation Genetic Testing of Aneuploidy and Segmental Imbalances / Jul 19, 2023